- やったのは以下。コード参照。
- そもそもフィルタリングと平滑化とは何だったか(関連: 状態空間モデルのまとめ絵 - クッキーの日記)。
- フィルタリング: 直接観測できない「状態」を、その時点の観測値までから推定。
- 平滑化: 直接観測できない「状態」を、すべての観測値を手に入れた後から推定。
- 以下の日足(直近250日)を拾ってきてつかった。R 内蔵のデータでなくわざわざこれをつかう意味はない。
- このデータに対してこれらのモデルが(そもそもDLMが)適当ということではない。実際にはちゃんと考えてモデルを決める。
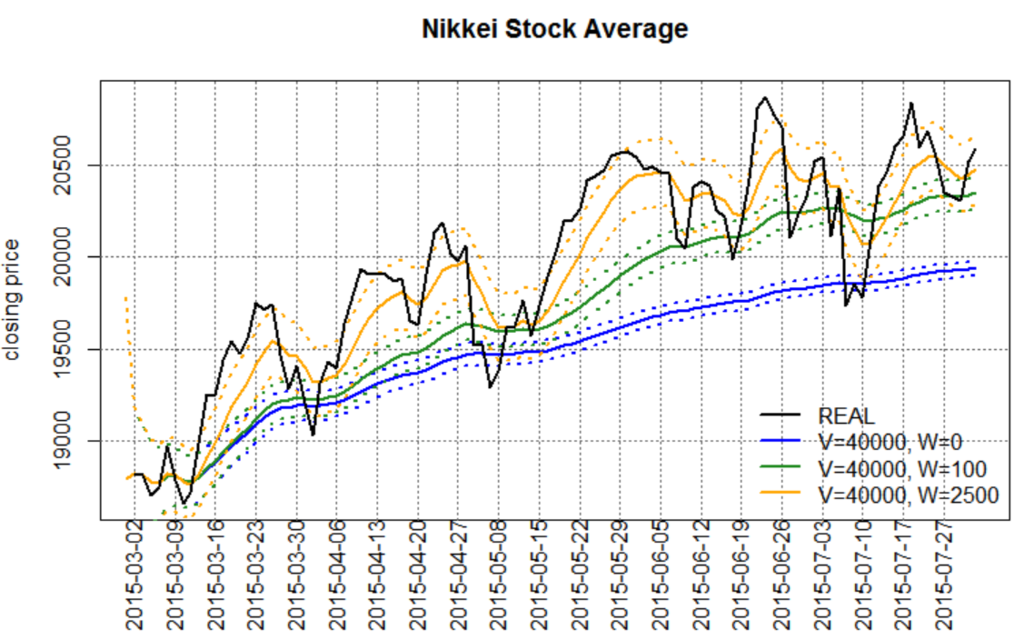
フィルタリング(ランダムウォーク・プラス・ノイズモデル)
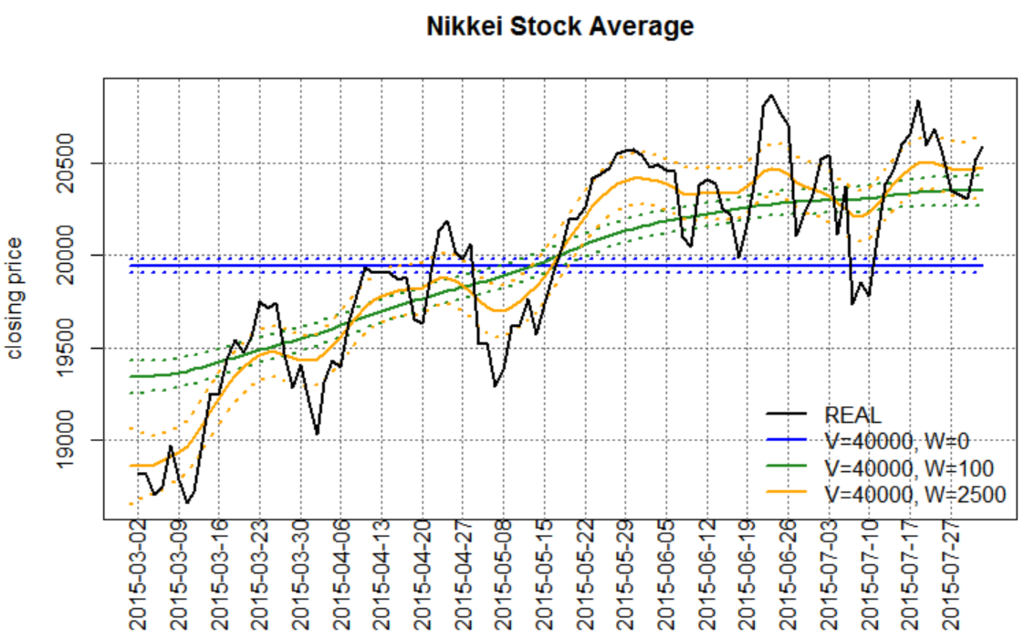
平滑化(ランダムウォーク・プラス・ノイズモデル)
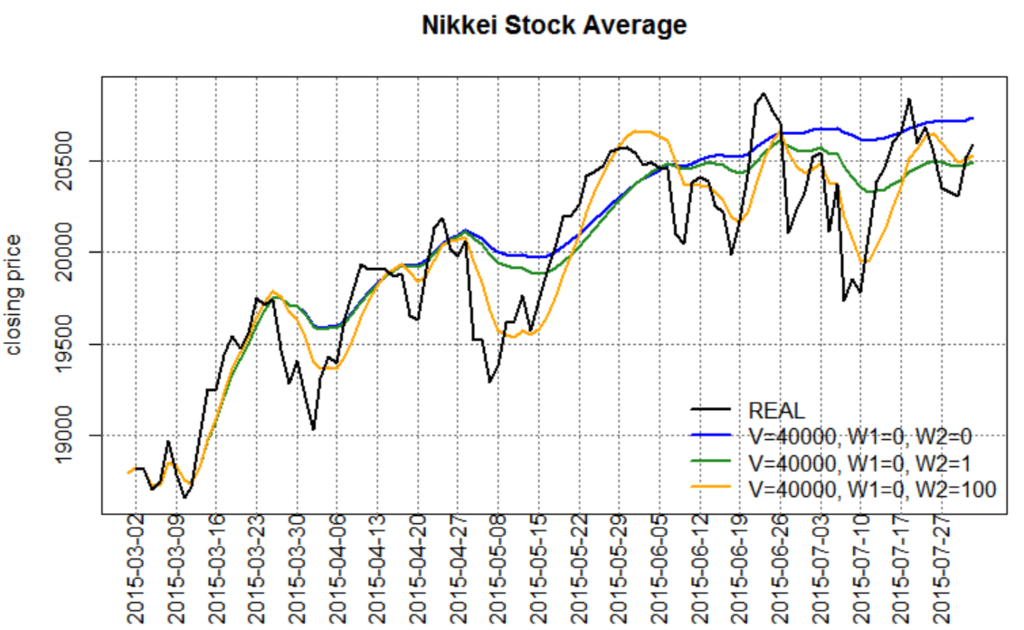
フィルタリング(線型成長モデル)
平滑化(線型成長モデル)
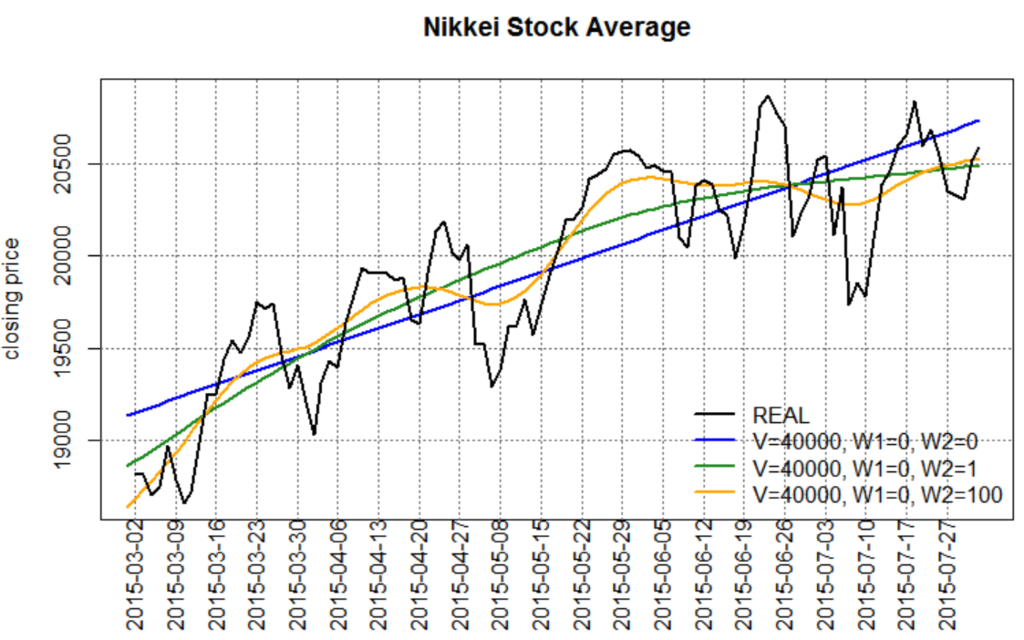
グラフの点線はフィルタ後/平滑化後の状態分布の95%点。
線型成長モデルについては、状態の平均には不確実性はなく(分散0)、状態の変化(成長)に不確実性がある(こちらに分散がある)と考えたモデルにしているので、グラフには分散をプロットしていない。
ランダムウォーク・プラス・ノイズモデルを描画するコードは以下(フィルタリングは dlmFilter の1行だけ)。
library(dlm) # データ読み込み nsa <- read.table("I101.csv", header=F, skip=2, sep=",", stringsAsFactors=FALSE) nsa <- nsa[rev(1:nrow(nsa)),] # 日付逆順のため colnames(nsa) <- c("date", "open", "high", "low", "close") nsa <- nsa[as.Date(nsa$date) >= as.Date("2015-03-01"),] # 絞り込み nsa <- nsa[as.Date(nsa$date) <= as.Date("2015-07-31"),] # 絞り込み # グラフ枠描画 plot(1:nrow(nsa), nsa$close, type="n", main="Nikkei Stock Average", xlab="", ylab="closing price", xaxt="n") indices <- seq(1, nrow(nsa), by=5) text(indices, par("usr")[3], labels=nsa$date[indices], srt=90, pos=2, offset=0, xpd=TRUE) abline(v=indices, h=seq(18000, 22000, by=500), col="dimgray", lty=3) # 凡例を溜めておく用 note <- data.frame(name="REAL", col="black", stringsAsFactors=FALSE) # ランダムウォーク・プラス・ノイズモデルのテンプレート rw.template <- dlm(FF=1, V=0, GG=1, W=0, m0=18797.94, C0=500*500) # 事前平均は2月終値 # 描画するグラフの切り替えフラグ flag.process <- "smoothing" # "filtering", "smoothing" # 描画用の便利関数(ランダムウォーク・プラス・ノイズモデル) add.plot.rw <- function(V, W, col) { rw <- rw.template V(rw) <- V W(rw) <- W nsaFilt <- dlmFilter(nsa$close, rw) # フィルタリング if (flag.process == "filtering") { # フィルタリングならフィルタ化分布を描画 lines(0:nrow(nsa), nsaFilt$m, lwd=2, col=col) sigma <- sqrt(unlist(dlmSvd2var(nsaFilt$U.C, nsaFilt$D.C))) # 各点のフィルタ後分散も描画 lines(0:nrow(nsa), nsaFilt$m + qnorm(0.025) * sigma, lwd=2, lty=3, col=col) lines(0:nrow(nsa), nsaFilt$m + qnorm(0.975) * sigma, lwd=2, lty=3, col=col) } else if (flag.process == "smoothing") { # 平滑化なら、さらに平滑化分布を描画 nsaSmooth <- dlmSmooth(nsaFilt) # 平滑化 lines(0:nrow(nsa), nsaSmooth$s, lwd=2, col=col) sigma <- sqrt(unlist(dlmSvd2var(nsaSmooth$U.S, nsaSmooth$D.S))) # 各点の平滑化後分散も描画 lines(0:nrow(nsa), nsaSmooth$s + qnorm(0.025) * sigma, lwd=2, lty=3, col=col) lines(0:nrow(nsa), nsaSmooth$s + qnorm(0.975) * sigma, lwd=2, lty=3, col=col) } note <- rbind(note, c(paste0("V=", V, ", W=", W), col)) return(note) } # 分散を色々変えて描く note <- add.plot.rw(V=40000, W=0, col="blue") note <- add.plot.rw(V=40000, W=100, col="forestgreen") note <- add.plot.rw(V=40000, W=2500, col="orange") # 元データと凡例 lines(1:nrow(nsa), nsa$close, lwd=2, col="black") legend("bottomright", legend=note$name, lwd=2, col=note$col, box.lwd=NA)
線型成長モデルの場合は該当箇所を以下に置き換える。
# 線型成長モデルのテンプレート lg.template <- dlm(FF=matrix(c(1, 0), nrow=1), V=0, GG=matrix(c(1, 0, 1, 1), nrow=2), W=0*diag(2), m0=c(18797.94, 0), C0=500*500*diag(2)) # 事前平均は2月終値 # 描画用の便利関数(線型成長モデル) add.plot.lg <- function(V, W1, W2, col) { lg <- lg.template V(lg) <- V W(lg)[1, 1] <- W1 W(lg)[2, 2] <- W2 nsaFilt <- dlmFilter(nsa$close, lg) # フィルタリング if (flag.process == "filtering") { # フィルタリングならフィルタ化分布を描画 lines(0:nrow(nsa), nsaFilt$m[,1], lwd=2, col=col) } else if (flag.process == "smoothing") { # 平滑化なら、さらに平滑化分布を描画 nsaSmooth <- dlmSmooth(nsaFilt) # 平滑化 lines(0:nrow(nsa), nsaSmooth$s[,1], lwd=2, col=col) } note <- rbind(note, c(paste0("V=", V, ", W1=", W1, ", W2=", W2), col)) return(note) } # 分散を色々変えて描く note <- add.plot.lg(V=40000, W1=0, W2=0, col="blue") note <- add.plot.lg(V=40000, W1=0, W2=1, col="forestgreen") note <- add.plot.lg(V=40000, W1=0, W2=100, col="orange")