Keras をよくわかっていないのでおかしな点があればご指摘いただきたいのですが、1つ前までの記事で紹介していた DAGMM(メモ1/メモ2/メモ3/メモ4)を Keras で実装しようとすると以下のような手順でできるんでしょうか。よくわかりません。
- 不整脈データ(274次元)を真ん中の特徴量が2次元の深層自己符号化器にかける。
- 深層自己符号化器が終わったところで、最初の入力と特徴量を回収してきて、損失に再構築誤差を流し込むと同時に、特徴量に再構築エラー(2次元)を concat してこれを出力する(カスタムレイヤー0)。
- さっきの出力を何層かの全結合層にかけて各分布への割り当て確率にする(不整脈データは混合数2)。
- ここで4次元の特徴量を回収してきて、尤度と正則化項を損失に流し込む(カスタムレイヤー1)。
ガワだけ実装すると以下のようなイメージですが、中身を実装してコンパイルできるのか知りませんし、訓練して勾配計算が意図通りに動くのかも知りません。
# DAGMM x_org = Input(shape=[274]) h_0 = Dense(10, activation='tanh')(x_org) z_c = Dense(2)(h_0) h_1 = Dense(10, activation='tanh')(z_c) x_dash = Dense(274)(h_1) z = MyLayer0()([x_org, z_c, x_dash]) # 損失に再構築誤差を付加 & 再構築エラーをconcat h_2 = Dense(10, activation='tanh')(z) h_3 = Dropout(0.5)(h_2) gamma = Dense(2, activation='softmax')(h_3) gamma_ = MyLayer1()([z, gamma]) # 損失にエネルギーと正則化を付加 DAGMM = Model(x_org, gamma_) DAGMM.compile(optimizer=optimizers.Adam(lr=0.0001), loss=None) DAGMM.summary()
_______________________________________________________________________________________
Layer (type) Output Shape Param # Connected to
=======================================================================================
input_1 (InputLayer) (None, 274) 0
_______________________________________________________________________________________
dense_1 (Dense) (None, 10) 2750 input_1[0][0]
_______________________________________________________________________________________
dense_2 (Dense) (None, 2) 22 dense_1[0][0]
_______________________________________________________________________________________
dense_3 (Dense) (None, 10) 30 dense_2[0][0]
_______________________________________________________________________________________
dense_4 (Dense) (None, 274) 3014 dense_3[0][0]
_______________________________________________________________________________________
my_layer0_1 (MyLayer0) (None, 4) 0 input_1[0][0]
dense_2[0][0]
dense_4[0][0]
_______________________________________________________________________________________
dense_5 (Dense) (None, 10) 50 my_layer0_1[0][0]
_______________________________________________________________________________________
dropout_1 (Dropout) (None, 10) 0 dense_5[0][0]
_______________________________________________________________________________________
dense_6 (Dense) (None, 2) 22 dropout_1[0][0]
_______________________________________________________________________________________
my_layer1_1 (MyLayer1) (None, 2) 0 my_layer0_1[0][0]
dense_6[0][0]
=======================================================================================
カスタムレイヤー0は以下です。再構築エラーをつくって低次元の特徴に concat すればいいと思うんですが、まだつくっていないのでなので低次元の特徴を2つ concat しています(不整脈データの例では低次元の特徴と再構築エラーが同じ2次元なので)。雰囲気です。
from keras import backend as K # 損失に再構築誤差を付加 & 再構築エラーをconcat class MyLayer0(Layer): def __init__(self, **kwargs): self.output_dim = 4 super(MyLayer0, self).__init__(**kwargs) def reconstruction_loss(self, x_org, x_dash): return metrics.mean_squared_error(x_org, x_dash) def reconstruction_error(self, x_org, x_dash): z_e = None # IMPLIMENT ME return z_e def call(self, inputs): x_org = inputs[0] z_c = inputs[1] x_dash = inputs[2] loss = self.reconstruction_loss(x_org, x_dash) self.add_loss(loss, inputs=[x_org, x_dash]) # 再構築誤差を付加 z_e = z_c # self.reconstruction_error(x_org, x_dash) # 再構築エラーを計算 z = K.concatenate([z_c, z_e], axis=1) return z def compute_output_shape(self, input_shape): return(input_shape[1][0], self.output_dim)
カスタムレイヤー1は以下です。ここは損失にエネルギー項と正則化項を足すだけですがまだ書いていません。一応各分布への割り当て gamma をそのまま output していますが output する必要はないです。
# 損失にエネルギーと正則化を付加 class MyLayer1(Layer): def __init__(self, **kwargs): self.output_dim = 2 super(MyLayer1, self).__init__(**kwargs) def custom_loss(self, z, gamma): return 0 # IMPLIMENT ME def call(self, inputs): z = inputs[0] gamma = inputs[1] loss = self.custom_loss(z, gamma) self.add_loss(loss, inputs=[z, gamma]) return gamma def compute_output_shape(self, input_shape): return(input_shape[1][0], self.output_dim)
まだ実装してないのでわかりませんというか実装すればいいんですが気が向いたらやります。いつ気が向くのかはわかりません。
深層自己符号化器までは何も問題ないはずなので取り出して動かしてみます。
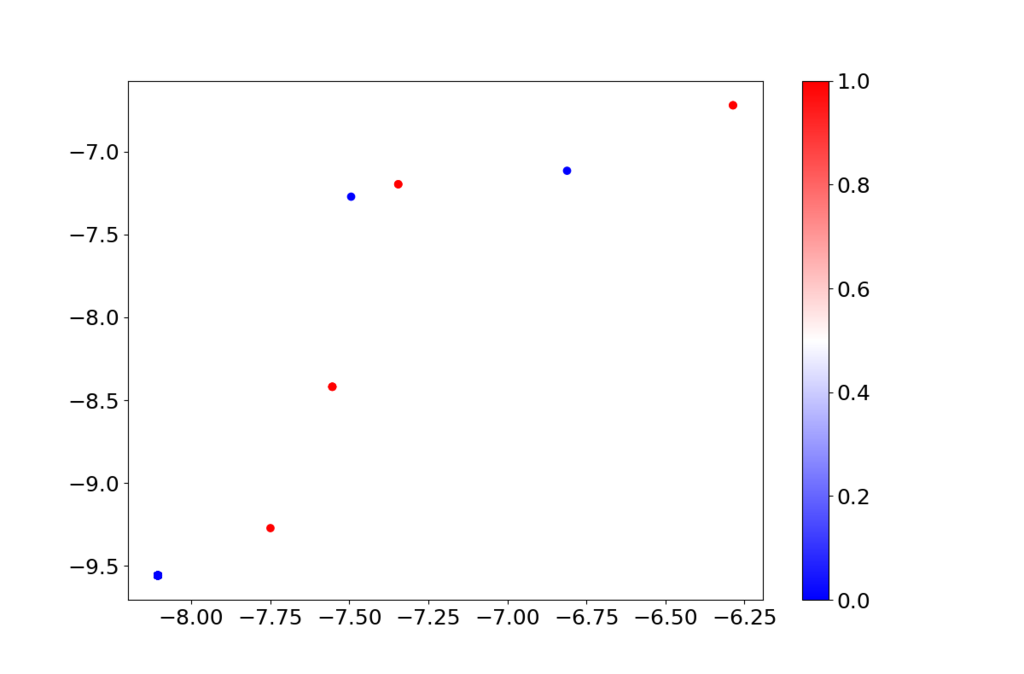
# 不整脈データ df = pd.read_csv('arrhythmia.data', header=None) df = df.drop(df.columns[[10,11,12,13,14]], axis=1) # 欠損カラムを除去 # 異常ラベルを貼り変える df.iloc[:,274][(df.iloc[:,274]>=1 ) & (df.iloc[:,274]<=2 )] = 20 df.iloc[:,274][(df.iloc[:,274]>=3 ) & (df.iloc[:,274]<=5 )] = 21 df.iloc[:,274][(df.iloc[:,274]>=6 ) & (df.iloc[:,274]<=6 )] = 20 df.iloc[:,274][(df.iloc[:,274]>=7 ) & (df.iloc[:,274]<=9 )] = 21 df.iloc[:,274][(df.iloc[:,274]>=10) & (df.iloc[:,274]<=13)] = 20 df.iloc[:,274][(df.iloc[:,274]>=14) & (df.iloc[:,274]<=15)] = 21 df.iloc[:,274][(df.iloc[:,274]>=16) & (df.iloc[:,274]<=16)] = 20 df.iloc[:,274] = df.iloc[:,274] - 20 x = df.drop(df.columns[[274]], axis=1).values y = df.iloc[:,274].values # 深層自己符号化器のみ取り出して学習 DAE = Model(x_org, x_dash) DAE.compile(loss='mean_squared_error', optimizer=optimizers.Adam(lr=0.0001)) DAE.fit(x, x, shuffle=True, epochs=10000, batch_size=128) # エンコーダを取り出してプロット encoder = Model(x_org, z_c) z_trained = encoder.predict(x) plt.figure(figsize=(12, 8)) plt.scatter(z_trained[:,0], z_trained[:,1], c=y, cmap=plt.get_cmap("bwr")) cb = plt.colorbar() for t in cb.ax.get_yticklabels(): t.set_fontsize(18) plt.tick_params(labelsize=18) plt.show()
上で学習すると特徴空間(2次元)は以下のようになりました。青が正常で赤が異常です。452点あったはずなんですが、相当重なっていると思います。
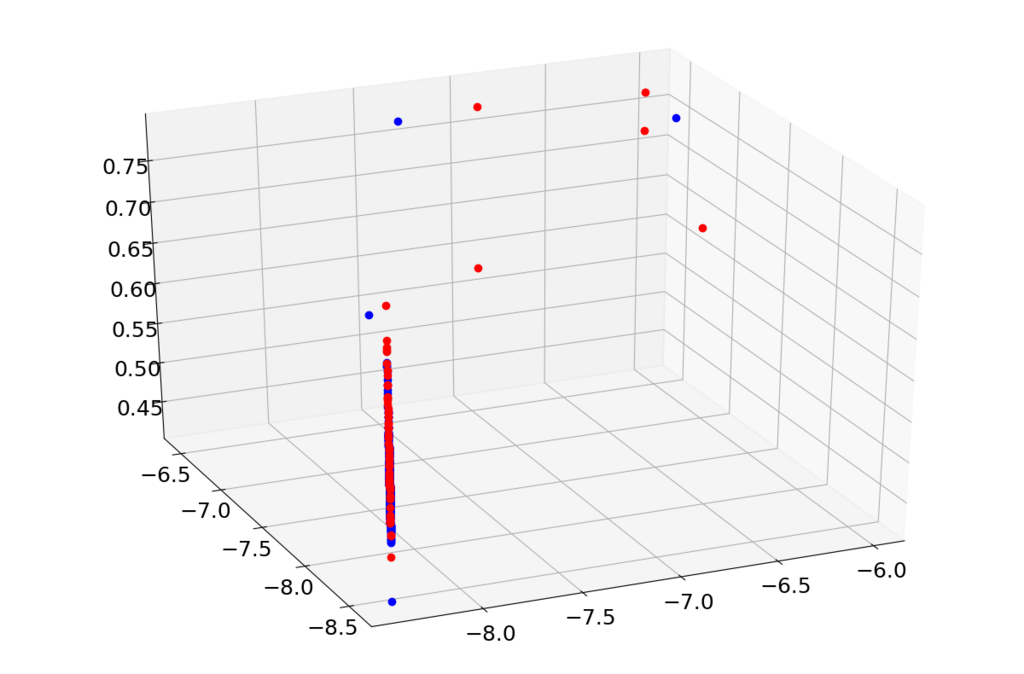
あと再構築誤差方向への分布はどうなっているのかも気になると思います。さらに何らかの再構築誤差も含めてプロットしようとするともう3次元プロットになってしまうのでその準備をします。
from mpl_toolkits.mplot3d import Axes3D def my_relative_distance(x, y): ret = [] for i in range(x.shape[0]): e2 = 0 x2 = 0 for j in range(x.shape[1]): e2 = e2 + (x[i][j] - y[i][j]) * (x[i][j] - y[i][j]) x2 = x2 + x[i][j] * x[i][j] ret.append(e2 / x2) return ret
その上でこうすると再構築誤差も含めてプロットできると思います。
# エンコーダを取り出してプロット encoder = Model(x_org, z_c) x0 = df.loc[df.iloc[:,274]==0,:].drop(df.columns[[274]], axis=1).values x1 = df.loc[df.iloc[:,274]==1,:].drop(df.columns[[274]], axis=1).values z_trained0 = encoder.predict(x0) z_trained1 = encoder.predict(x1) r_error0 = my_relative_distance(x0, DAE.predict(x0)) r_error1 = my_relative_distance(x1, DAE.predict(x1)) fig = plt.figure(figsize=(12, 8)) ax = Axes3D(fig) ax.plot(z_trained0[:,0], z_trained0[:,1], r_error0, "o", color="#0000ff") ax.plot(z_trained1[:,0], z_trained1[:,1], r_error1, "o", color="#ff0000") plt.tick_params(labelsize=18) plt.show()
なんか再構築誤差方向にも異常データと正常データがあまり分離されているようにみえないので、深層自己符号化器のみで分離する場合は適するハイパーパラメータが違うんだと思います。